파이썬 기본특성
✅ 최신버전사용 주의 : 서로 버전 충돌 발생하는 경우가 있기 때문에, 파이썬 오픈소스 라이브러리 사용시, 최신버전 사용은 가급적 지양할 필요가 있다.
✅ 파이썬 변수 데이터 구조 : 파이썬은 '참조기반구조' 자료구조형태로, 예를 들어 A = 100 라는 코드는 타 언어의 경우, A라는 정해진 공간을 확보하지만, 파이썬은 정해지지 않은 넉넉한 메모리 별도 공간에 id주소를 매겨서 100의 값을 넣어 둔 후, 그 값을 A변수로 호출해 온다는 의미가 된다. 따라서 A변수에는 데이터 id 주소가 할당되어 있다. 다시 말해, A는 데이터 공간이 아니라, 단지 다른 넓은 데이터 저장공간에서 해당 id주소에 들어간 데이터를 불러와서 가지고 있는 그릇, 즉 참조변수이다. list_a = [1,2,3,4,5]를 예를 들면, 타 언어는 리스트 [ ] 안에 동일한 속성값(숫자, integer)를 넣어야 한다. 그러나 파이썬은 [ ] 안에 서로 다른 속성값을 list_a = [1,2,3,4,'abc'] 이와같이 넣어도 저장이 가능하다. 왜냐하면 파이썬은 데이터를 별도의 넓은 공간에 저장하여 불러오기 (참조하기) 때문에 어떤 데이터 속성도 변수에 넣는 것이 가능하다.
✅ copy()함수 : 파이썬 변수는 값을 가지고 있는 것이 아니라, 별도의 넓은 공간에 들어 있는 값의 id값을 참조하여 가지고 있기 때문에, id가 아닌 변수가 참조하는 값만 복사하려면 copy()함수를 사용한다. 즉, B = A.copy() 이렇게 하면 B에는 다른 id 주소에 100이라는 값이 할당되어 참조된다. 반면 B = A 이어도 역시 B에는 100이라는 값이 있다. 그러나 B는 A변수가 가지고 있는 id와 동일한 id를 갖게 되어, 나중에 B의 값을 변경하게 되면, A값도 따라서 변경되게 된다. 그래서 원본값을 보존하게 위해 다른 id로 값을 복사해 넣는 기능이 바로 copy()인 것이다.
✅ 연산 : 리스트는 리스트간 연산은 불가능하다. 그러나 넘파이의 행렬은 행렬간 연산이 가능하다.
input() : 숫자를 입력받아도 문자로 입력받는다.
Data ---> Feature Extraction ----> Modelling -----> Output
Rule base | <---------------------Human------------------> |
ML | <---------Human---------> | <--Machine--> |
DL | <------------------Machine -------------------> |
- Machine Learning : 지도학습, 비지도학습, 강화학습
- 지도학습 : 분류, 회귀
- 비지도학습 : 군집, Feature Extraction, Model Generation
- 강화학습 : 현재 상태에서 어떤 행동을 선택해야 가장 이득이 되는가에 대한 학습, Trial & error를 통한 학습, 최적화와 관련 있는 학습
- Linear Regression
- Scikit-learn
- 모델 생성 ▷ 모델 학습 ▷ 예측
데이터가 어떻게 생겼는지 알 수가 있다. = 데이터의 분포를 이해할 수가 있다.
머신러닝은 궁극적으로 적용성이 가장 문제이다. 그 적용분야에 어떤 통계기법을 사용하는가에 대한 판단이 중요하다.
지도학습, 비지도학습, 강화학습의 적용에 대한 판단을 현업 성격에 맞게 사용해야 한다.
파이썬
- 반복문
for i in [1,2,3,4,5,6,7]:
print(i)인덱스로 요소를 불러와서 반복문을 돌리는 C언어와는 달리 '리스트'를 이용하여 요소값 자체를 바로 불러와서 반복문을 실행할 수가 있다. 인덱스로 리스트 요소를 불러오기 위해서는 range()를 사용한다.
for i in range(1,10,3):
print(i)※ range(a,b,c) : a~b까지 c간격으로 숫자 가져오기
while (조건):
반복문 수행
i = 1
while s < 100:
s = s + i
i = i + 1
s, i = 0, 1
for i in range(1,100):
s = s + i
if s > 100 : break가급적이면 처음에는 for문으로 코딩하고 그 다음에 익숙해 졌을 때, while문을 사용하여 코딩한다. 위 while문과 for문은 같은 작업을 수행한다. 즉, for문으로 작성할 수 있는 것을 while문으로 만들 수 있고 while문으로 작성한 것을 for문으로 만들 수가 있다.
- 리스트 : read and write 모두 가능, 리스트 내 값을 바꿀 수가 있음
- 튜플 : read만 가능, 튜플에 값을 입력하면 값을 바꿀 수가 없다, 읽어 오기만 함
- 딕셔너리
st_index = ['학번', '이름', '학과']
st_info = ['1000', '김보경', '파이썬학과']
st_dict = {'학번':'1000', '이름':'김보경', '학과':'파이썬학과'}※ Dictionary → {key:value}로 구성되어 있음
- 함수
- 개념 : 붕어빵틀
- 반복적인 코드를 함수라는 형태로 만들어 놓음 : 반복 코드작업을 줄이고자 하는 목적
s,a,b = 0, 10,20
for i in range(a, b+1):
s += i
print(s)
# 함수생성
def summation(a,b):
# summation : a~b까지의 합
s = 0
for i in range(a, b+1):
s += i
return s - 함수 사용 목적 : 코드 협업
- 함수 내부의 내용을 건드리지 않기 때문에 협업 효율 증가
- 클래스
- 개념 : 함수 여러개를 묶어 놓은 것
- 객체 (object) => themes 와 method
[선형대수]
- 벡터 : 크기와 방향을 가지는 물리량
- 모든 데이터는 컴퓨터에 들어갈 때, 벡터로 표현되어지게 되어 있음, 즉 모든 데이터는 크기와 방향으로 표현해야 함.
강아지 : 0 ==> 0,1,2
고양이 : 1 ==> 1,0,2
팬더 : 2 ==> 2,1,0
- one hot encoding
강아지 고양이 팬더
1 0 0
0 1 0
0 0 1
3가지 요소가 공평한 거리, x,y,z의 방향으로 표현할 수 있기 때문에 좋은 데이터 변환임
- 유클리디안 거리
두 벡터 사이의 거리 = 두 데이터간 유사성(Similarity)을 말해 줌, 데이터들의 유사도를 측정할 수 있는 척도중의 하나임. 결국 feature extraction은 유사성 있는 것끼리 묶어서 표현하는 것과 같음
- 고유벡터
- mapping : 갖고 있는 데이터에 '특정 행렬'을 곱하여 다른 방향의 축에서 데이터 특성을 볼 수 있게 하는 작업으로 데이터의 feature extraction을 위해서 하는 작업임
[확률이론]
확률과 통계이론의 원리 및 적용 : 데이터가 충분히 모이게 되면 관통하는 유의미한 특성이 나타난다는 것. 모아놓은 데이터를 잘 분석해 놓았다가 새로운 데이터가 들어오는 이벤트가 발생하였을 때, 그 데이터의 특성을 맞추는 것, 과거의 데이터처럼 미래에도 똑같이 형태의 데이터가 계속 들어올 것이라는 가정.
- bias : 데이터의 편향정도를 나타냄
- bias와 분산
- 공분산 : 데이터의 분산 정도에 따른 상관관계를 보는 척도
- 조건부 확률 : 베이즈의 정리
- P[B|A] : 어떤 데이터(A)가 들어 왔다는 가정 하에 특정값(B)을 뽑아내는 것
- P[A'|B] P[B] / P[A'] : 반대로 그 데이터(A)에서 특정값(B)을 뽑아서 확률분포 특성정의한 상태(지도학습)에서 새로 들어온 데이터(A')의 특성을 맞추게 됨 → 특정값 B를 가지고 표현했기 때문에, 지도학습이 전제가 된 상태임
- Likelihood : 가장 가능성 높음
- Maximum Likelihood Estimator
[선형회귀]
선형회귀분석 : Least mean square을 최소화해 주는 직선을 찾는 분석
y = ax + a2x1 +...
Pseudo Inverse
scikit-learn
BSD license
[선형회귀모델 만들기 순서]
1. 원하는 ML 모델을 만든다.
2. fit 이라는 method 함수로 학습을 한다.
3. predict라고 하는 method 함수로 예측을 한다.
import numpy as np
from sklearn.linear_model import LinearRegression
X = np.array([[1, 1], [1, 2], [2, 2], [2, 3]])
# y = 1 * x_0 + 2 * x_1 + 3
y = np.dot(X, np.array([1, 2])) + 3
# 1) 머신러닝 모델 만들기
reg = LinearRegression(fit_intercept = False)
# 2) fit이라는 method로 학습
reg.fit(X,y)
# 3) Predict 명령어로 예측
x_pred = reg.predict(X)
print(x_pred)
reg.predict([[11,12]])
print(reg.score(X, y),
reg.coef_ ,
reg.intercept_,
reg.predict(np.array([[3, 5]])))
- LinearRegression(*,fit_intercept=True, normalize='deprecated', copy_X=True, n_jobs=None, positive=False)
import matplotlib.pyplot as plt
import numpy as np
from sklearn import datasets, linear_model
from sklearn.metrics import mean_squared_error, r2_score
# Load the diabetes dataset
diabetes = datasets.load_diabetes()
# Use only one feature
diabetes_X = diabetes.data[:, np.newaxis, 2]
# Split the data into training/testing sets
diabetes_X_train = diabetes_X[:-20]
diabetes_X_test = diabetes_X[-20:]
# Split the targets into training/testing sets
diabetes_y_train = diabetes.target[:-20]
diabetes_y_test = diabetes.target[-20:]
# Create linear regression object : fill here
regr = linear_model.LinearRegression()
# Train the model using the training sets : fill here
regr.fit(diabetes_X_train,diabetes_y_train)
# Make predictions using the testing set : fill here
diabetes_y_pred = regr.predict(diabetes_X_test)
# The coefficients
print('Coefficients: \n', regr.coef_)
# The mean squared error
print("Mean squared error: %.2f"
% mean_squared_error(diabetes_y_test, diabetes_y_pred))
# Explained variance score: 1 is perfect prediction
print('Variance score: %.2f' % r2_score(diabetes_y_test, diabetes_y_pred))
# Plot outputs
plt.scatter(diabetes_X_test, diabetes_y_test, color='black')
plt.plot(diabetes_X_test, diabetes_y_pred, color='blue', linewidth=3)
plt.xticks(())
plt.yticks(())
plt.show()Coefficients:
[938.23786125]
Mean squared error: 2548.07
Variance score: 0.47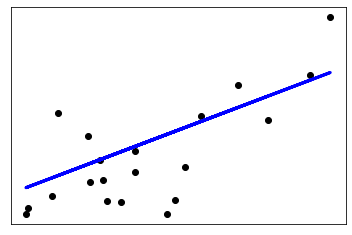
- Iris 데이터를 이용한 선형회귀모델 생성
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.linear_model import LinearRegression
iris = datasets.load_iris()
X = iris.data
y = iris.target
# 1) ML 모델생성
mdl = LinearRegression()
# 2) Fit으로 학습
mdl.fit(X,y)
# 3) 예측
y_pred = mdl.predict(X)
print(reg.score(X, y), reg.coef_ , reg.intercept_)
print('Coefficients: \n', reg.coef_)
print("Mean squared error: %.2f" % mean_squared_error(y, y_pred))
# Explained variance score: 1 is perfect prediction
print('Variance score: %.2f' % r2_score(y, y_pred))
print(accuracy_score(y,y_pred))0.9303939218549563 [-0.11190585 -0.04007949 0.22864503 0.60925205] 0.18649524720624933
Coefficients:
[-0.11190585 -0.04007949 0.22864503 0.60925205]
Mean squared error: 0.03
Variance score: 0.96
0.9733333333333334
- accuracy_score 확인
error = np.abs(y-y_pred)
print(error)
print(np.nonzero(error))
print(np.nonzero(error)[0])
print(np.nonzero(error)[0].shape)
acc = 1 - np.nonzero(error)[0].shape[0] / y.shape[0]
print(acc)[0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1.
0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0. 1. 0. 0. 0. 0. 0. 0. 0. 0. 0. 0.
0. 0. 0. 0. 0. 0.]
(array([ 70, 83, 119, 133], dtype=int64),)
[ 70 83 119 133]
(4,)
0.9733333333333334- 위 분석은 전 데이터를 가지고 모델을 만들었기 때문에 정확하지 않다. 그래서 데이터를 학습데이터, 검증데이터로 1/3 나눠서 모델생성하고 정확도 확인
import numpy as np
from sklearn import datasets
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error, r2_score, accuracy_score
from sklearn.model_selection import train_test_split
iris = datasets.load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=1/3)
reg = LinearRegression()
reg.fit(X_train, y_train)
y_pred = np.round(reg.predict(X_test))
print('Coefficients: \n', reg.coef_)
print("Mean squared error: %.2f" % mean_squared_error(y_test, y_pred))
print('Variance score: %.3f' % r2_score(y_test, y_pred))
print('accuracy_score: %.3f' % accuracy_score(y_test,y_pred))0.9296795792958372 [-0.09314565 -0.01430974 0.21616868 0.64395766] 0.00779856567884929
Coefficients:
[-0.09314565 -0.01430974 0.21616868 0.64395766]
Mean squared error: 0.04
Variance score: 0.937
accuracy_score: 0.960
- 와인데이터를 이용한 선형회귀모델 생성
import numpy as np
from sklearn import datasets
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error, r2_score, accuracy_score
from sklearn.model_selection import train_test_split
X, y = wine.data, wine.target
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=1/4)
reg = LinearRegression()
reg.fit(X_train,y_train)
y_pred = np.round(reg.predict(X_test))
print('* Score :', reg.score(X, y))
print('* Coefficients :\n', reg.coef_)
print('* Intercept :', reg.intercept_)
print('* Mean squared error: %.2f' % mean_squared_error(y_test, y_pred))
print('* Variance score : %.3f' % r2_score(y_test, y_pred))
print('* accuracy_score : %.3f' % accuracy_score(y_test,y_pred))* Score : 0.89759554249323
* Coefficients :
[-0.1195491 0.03419282 -0.13126391 0.03335756 -0.00088314 0.07815756
-0.3294019 -0.26355738 0.03745609 0.09028364 -0.1975514 -0.24512435
-0.0007623 ]
* Intercept : 3.6344324928929614
* Mean squared error: 0.07
* Variance score : 0.896
* accuracy_score : 0.933
- Overfitting vs Underfitting
import numpy as np
import matplotlib.pyplot as plt
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import PolynomialFeatures
from sklearn.linear_model import LinearRegression
from sklearn.model_selection import cross_val_score
def true_fun(X):
return np.cos(1.5 * np.pi * X)
np.random.seed(0)
n_samples = 30
degrees = [1, 4, 15, 30]
X = np.sort(np.random.rand(n_samples))
y = true_fun(X) + np.random.randn(n_samples) * 0.1
plt.figure(figsize=(14, 5))
for i in range(len(degrees)):
ax = plt.subplot(1, len(degrees), i + 1)
plt.setp(ax, xticks=(), yticks=())
polynomial_features = PolynomialFeatures(degree=degrees[i],
include_bias=False)
linear_regression = LinearRegression()
pipeline = Pipeline([("polynomial_features", polynomial_features),
("linear_regression", linear_regression)])
pipeline.fit(X[:, np.newaxis], y)
# Evaluate the models using crossvalidation
scores = cross_val_score(pipeline, X[:, np.newaxis], y,
scoring="neg_mean_squared_error", cv=10)
X_test = np.linspace(0, 1, 100)
plt.plot(X_test, pipeline.predict(X_test[:, np.newaxis]), label="Model")
plt.plot(X_test, true_fun(X_test), label="True function")
plt.scatter(X, y, edgecolor='b', s=20, label="Samples")
plt.xlabel("x")
plt.ylabel("y")
plt.xlim((0, 1))
plt.ylim((-2, 2))
plt.legend(loc="best")
plt.title("Degree {}\nMSE = {:.2e}(+/- {:.2e})".format(
degrees[i], -scores.mean(), scores.std()))
plt.show()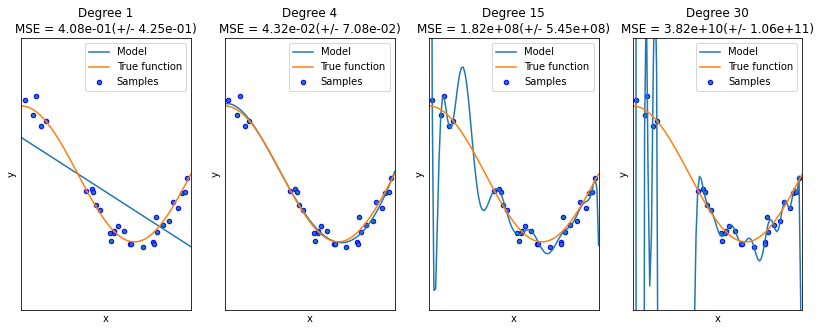
[벡터 양자화]
- Clustering (군집) : N개 특징 벡터들의 집합을 N개 특징 벡터들의 집합으로 mapping시키는 것
- k-means clustering : 군집의 중심을 기준으로
<Logic>
0) 랜덤으로 선택 시작, 군집 갯수 N 선택 필요, 데이터 속에 중심을 랜덤하게 잡는다.
1) 군집의 중심 구하기 : 특정 군집의 데이터 간 평균을 구함
2) 군집 분류는 각각의 군집 중심에서 가장 가까운 군집에 속하는 곳으로 소속
※ 1)과 2)이 동작하게 하기 위해서는 반드시 0)이 전제되어야 함.
시작
E단계
M단계
왜곡 판단
- 실루엣 파라미터 Silhouette Parameter
군집이 잘 시작되었는지 군집 갯수가 잘 선택되었는지 알 수가 없기 때문에 최소한의 기준을 세우기 위해서 실루엣 파라미터가 필요하다.
# Generating the sample data from make_blobs
# This particular setting has one distinct cluster and 3 clusters placed close
# together.
X, y = make_blobs(n_samples=500,
n_features=2,
centers=4,
cluster_std=1,
center_box=(-10.0, 10.0),
shuffle=True,
random_state=1)
* n_samples : 데이터 샘플 갯수
* n_features : n개의 차원
* centers : 중심 갯수
* cluster_std : 클러스터가 퍼져있는 정도
* center_box : 박스 전체 좌표
* shuffle
* random_state : 재현 가능성 (※ random seed)
clusterer = KMeans(n_clusters=n_clusters, random_state=0)
cluster_labels = clusterer.fit_predict(X)
# The silhouette_score gives the average value for all the samples.
# This gives a perspective into the density and separation of the formed
# clusters
silhouette_avg = silhouette_score(X, cluster_labels)
[실루엣 평가값 의미]
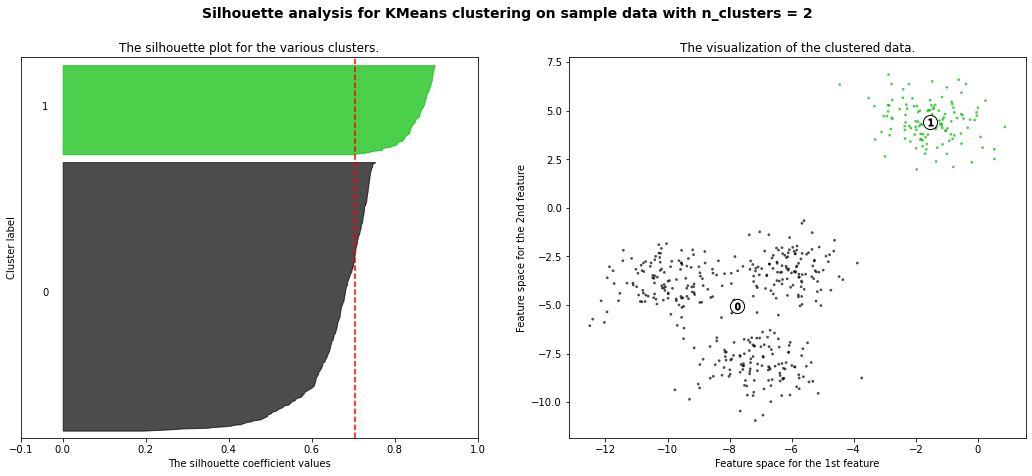
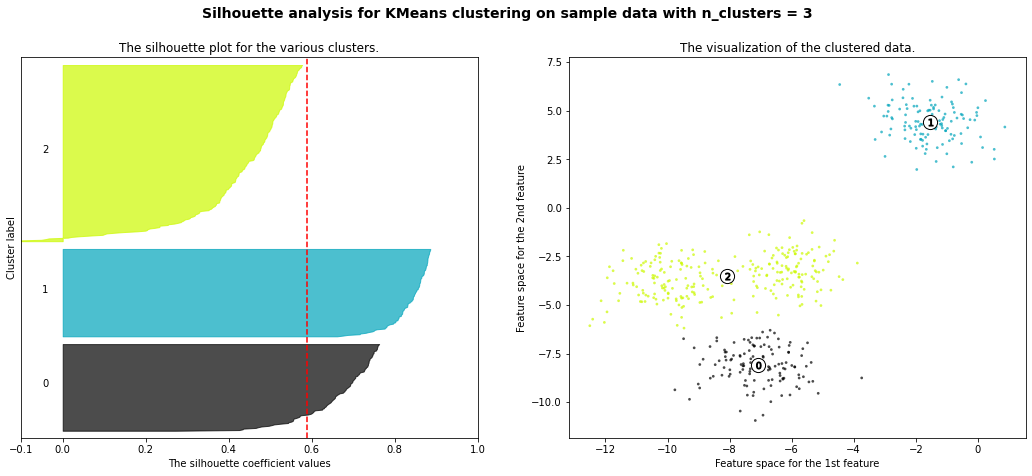
- 잘 됨 : 클러스터 중심값에 가깝고 나머지 중심값에서 멀면 1에 가까움
- 안 됨 : 클러스터 중심값에도 가깝고 나머지 중심값에도 가까우면 0으로 가면서 최종 -1로 감
- 안 됨 : 클러스터 중심값에도 멀고 나머지 중심값에서도 멀면 0으로 가면서 최종 -1로 감
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
from sklearn.metrics import silhouette_samples, silhouette_score
import matplotlib.pyplot as plt
import matplotlib.cm as cm
import numpy as np
# Generating the sample data from make_blobs
# This particular setting has one distinct cluster and 3 clusters placed close
# together.
X, y = make_blobs(n_samples=500,
n_features=2,
centers=4,
cluster_std=1,
center_box=(-10.0, 10.0),
shuffle=True,
random_state=1) # For reproducibility
range_n_clusters = [2, 3, 4, 5, 6]
for n_clusters in range_n_clusters:
# Create a subplot with 1 row and 2 columns
fig, (ax1, ax2) = plt.subplots(1, 2)
fig.set_size_inches(18, 7)
# The 1st subplot is the silhouette plot
# The silhouette coefficient can range from -1, 1 but in this example all
# lie within [-0.1, 1]
ax1.set_xlim([-0.1, 1])
# The (n_clusters+1)*10 is for inserting blank space between silhouette
# plots of individual clusters, to demarcate them clearly.
ax1.set_ylim([0, len(X) + (n_clusters + 1) * 10])
# Initialize the clusterer with n_clusters value and a random generator
# seed of 10 for reproducibility.
clusterer = KMeans(n_clusters=n_clusters, random_state=0)
cluster_labels = clusterer.fit_predict(X)
# The silhouette_score gives the average value for all the samples.
# This gives a perspective into the density and separation of the formed
# clusters
silhouette_avg = silhouette_score(X, cluster_labels)
print("For n_clusters =", n_clusters,
"The average silhouette_score is :", silhouette_avg)
# Compute the silhouette scores for each sample
sample_silhouette_values = silhouette_samples(X, cluster_labels)
y_lower = 10
for i in range(n_clusters):
# Aggregate the silhouette scores for samples belonging to
# cluster i, and sort them
ith_cluster_silhouette_values = \
sample_silhouette_values[cluster_labels == i]
ith_cluster_silhouette_values.sort()
size_cluster_i = ith_cluster_silhouette_values.shape[0]
y_upper = y_lower + size_cluster_i
color = cm.nipy_spectral(float(i) / n_clusters)
ax1.fill_betweenx(np.arange(y_lower, y_upper),
0, ith_cluster_silhouette_values,
facecolor=color, edgecolor=color, alpha=0.7)
# Label the silhouette plots with their cluster numbers at the middle
ax1.text(-0.05, y_lower + 0.5 * size_cluster_i, str(i))
# Compute the new y_lower for next plot
y_lower = y_upper + 10 # 10 for the 0 samples
ax1.set_title("The silhouette plot for the various clusters.")
ax1.set_xlabel("The silhouette coefficient values")
ax1.set_ylabel("Cluster label")
# The vertical line for average silhouette score of all the values
ax1.axvline(x=silhouette_avg, color="red", linestyle="--")
ax1.set_yticks([]) # Clear the yaxis labels / ticks
ax1.set_xticks([-0.1, 0, 0.2, 0.4, 0.6, 0.8, 1])
# 2nd Plot showing the actual clusters formed
colors = cm.nipy_spectral(cluster_labels.astype(float) / n_clusters)
ax2.scatter(X[:, 0], X[:, 1], marker='.', s=30, lw=0, alpha=0.7,
c=colors, edgecolor='k')
# Labeling the clusters
centers = clusterer.cluster_centers_
# Draw white circles at cluster centers
ax2.scatter(centers[:, 0], centers[:, 1], marker='o',
c="white", alpha=1, s=200, edgecolor='k')
for i, c in enumerate(centers):
ax2.scatter(c[0], c[1], marker='$%d$' % i, alpha=1,
s=50, edgecolor='k')
ax2.set_title("The visualization of the clustered data.")
ax2.set_xlabel("Feature space for the 1st feature")
ax2.set_ylabel("Feature space for the 2nd feature")
plt.suptitle(("Silhouette analysis for KMeans clustering on sample data "
"with n_clusters = %d" % n_clusters),
fontsize=14, fontweight='bold')
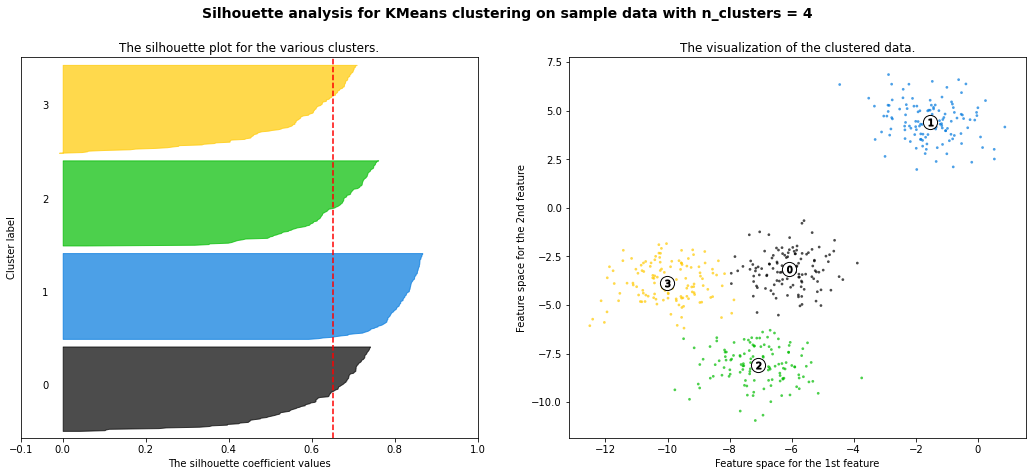
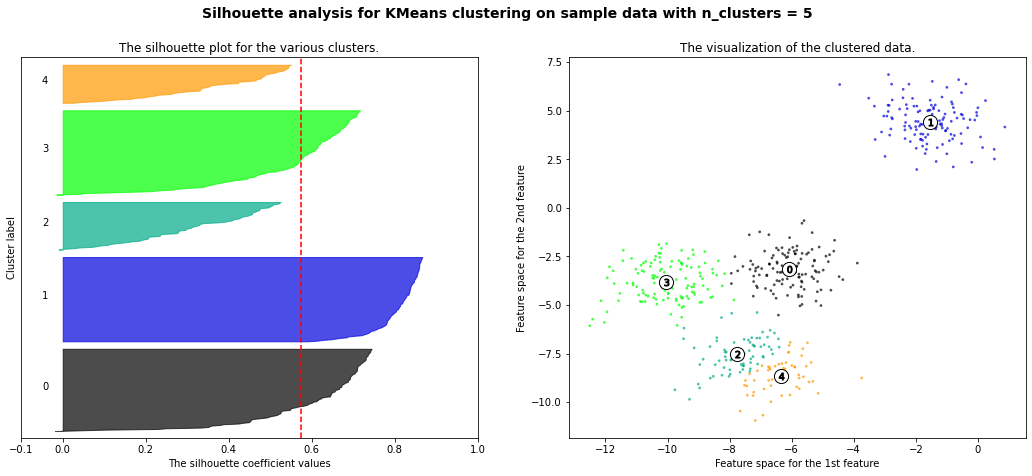
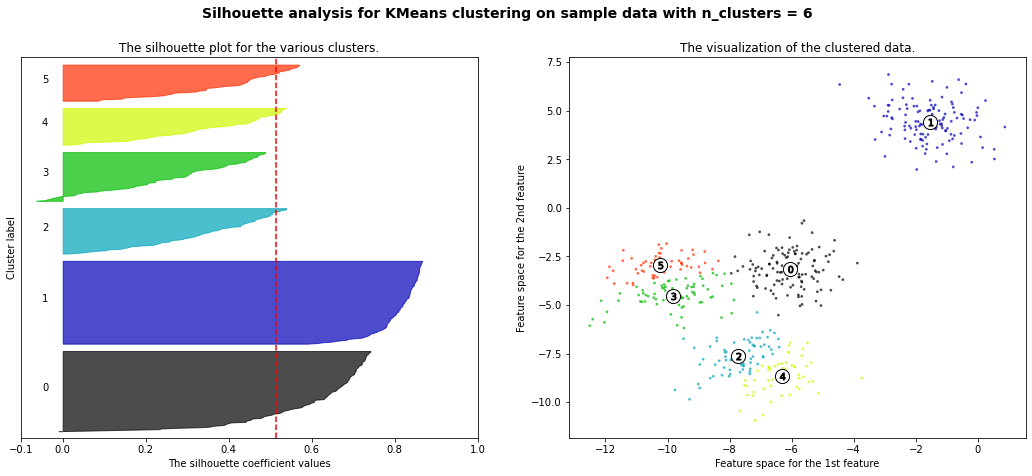
plt.show()For n_clusters = 2 The average silhouette_score is : 0.7049787496083261
For n_clusters = 3 The average silhouette_score is : 0.5882004012129721
For n_clusters = 4 The average silhouette_score is : 0.6505186632729437
For n_clusters = 5 The average silhouette_score is : 0.5746932321727456
For n_clusters = 6 The average silhouette_score is : 0.5150064498560357
[Principle Component Analysis]
scaling
unequal scaling
rotation
horizontal shear
hyperbolic rotation
원하는 형태로 데이터를 바라보고자 데이터 벡터 차원을 mapping 해주는 것, 보통 차원 축소용으로 사용됨
데이터를 싸고 있는 알맹이만 보면 되는데 껍데기까지 데이터를 분석하면 분석 정확도가 떨어짐
데이터를 판단하는데에 중요한 핵심 인자만 분석하여 사용하겠다는 것
PCA : 데이터가 가장 잘 보이는 축을 찾는 것,
- 데이터가 가장 잘 보이는 축 = 데이터의 공분산이 가장 큰 축
※ 공분산 (covariance) : 두 개의 확률 변수의 흩어진 정도, X와 Y의 각각의 편차를 곱한 것의 평균
Cov(X, Y) > 0 : X가 증가 할 때 Y도 증가한다.
Cov(X, Y) < 0 : X가 증가 할 때 Y는 감소한다.
Cov(X, Y) = 0
: 공분산이 0이라면 두 변수간에는 아무런 선형관계가 없으며
두 변수는 서로 독립적인 관계에 있음을 알 수 있다.
그러나 두 변수가 독립적이라면 공분산은 0이 되지만,
공분산이 0이라고 해서 항상 독립적이라고 할 수 없다.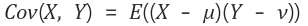
[PCA()와 pca.fit()까지 하는 일]
1. 공분산 행렬
2. 고유분석
3. M개의 가장 큰 고유값 선택
4. 선택된 고유값과 관련된 고유벡터를 구하고 연결하여 변환행렬 W 만듬
import numpy as np
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
X = np.array([[-1, -1], [-2, -1], [-3, -2], [1, 1], [2, 1], [3, 2]])
# 모델 생성
pca = PCA(n_components=2)
# fit으로 학습
pca.fit(X)
plt.scatter(X[:,0], X[:,1])
# X값을 PCA분석된 값으로 변환
x_trans = pca.transform(X)
plt.scatter(x_trans[:,0], x_trans[:,1])
print(pca.singular_values_)
print(pca.explained_variance_ratio_)[6.30061232 0.54980396]
[0.99244289 0.00755711]

y축 기준 공분산 고유값 : 6.300, x축 기준 공분산 고유값 : 0.549
y축에서 흩어짐 정도 : 0.992 (매우 높음)
x축에서 흩어짐 정도 : 0.007 (매우 낮음)
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import load_iris
from sklearn import decomposition
from sklearn.decomposition import PCA
from sklearn.linear_model import LinearRegression
from sklearn.metrics import accuracy_score
from sklearn.model_selection import train_test_split
# 1) IRIS 데이터셋을 들고온다
iris = datasets.load_iris()
X = iris.data
y = iris.target
# 2) Train / Test split을 한다
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=1/3)
# 3) Train 데이터를 PCA를 통해 적당히 차원을 축소한다.
pca = PCA(n_components=2)
pca.fit(X_train)
X_train_pca = pca.transform(X_train)
X_test_pca = pca.transform(X_test)
print(pca.singular_values_)
print(pca.explained_variance_ratio_)
plt.scatter(X_train_pca[:,0], X_train_pca[:,1])
plt.scatter(X_test_pca[:,0], X_test_pca[:,1])[20.91043539 4.95587209]
[0.92828191 0.05214275]
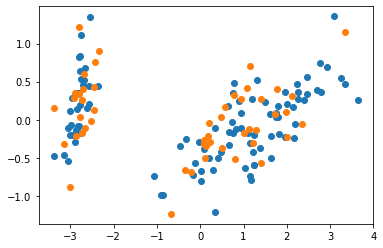
# 4) PCA를 통한 데이터로 LR 모델을 학습
mdl1 = LinearRegression()
mdl1.fit(X_train, y_train)
mdl2 = LinearRegression()
mdl2.fit(X_train_pca, y_train)
# 5) LR 모델로 예측하여 성능을 검토
y_pred1 = np.abs(np.round(mdl1.predict(X_test)))
y_pred2 = np.abs(np.round(mdl2.predict(X_test_pca)))
print('원데이터 회귀모델성능 : ', accuracy_score(y_test, y_pred1))
print('PCA데이터 회귀모델성능 : ', accuracy_score(y_test, y_pred2))원데이터 회귀모델성능 : 0.96
PCA데이터 회귀모델성능 : 0.96
[Linear Discriminant Analysis] 선형판별분석
LDA는 데이터를 가장 잘 나눠주는 축을 찾는 것
1) PCA로 데이터가 가장 잘 분산되어 있는 방향으로 데이터를 보고
2) LDA로 그 분산되어 있는 데이터를 잘 분류하는 축을 찾아주는 것임
두 데이터 사이의
1. 평균이 멀어지고 (다른 종류의 데이터는 최대한 멀리 떨어져 있어야 함) ▷ 최대값
2. 데이터 집단 자체의 분산은 작아지게 하고 (같은 종류의 데이터는 최대한 모여져 있고) ▷ 최소값
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn import decomposition
from sklearn.decomposition import PCA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
from sklearn.linear_model import LinearRegression
from sklearn.metrics import mean_squared_error, r2_score, accuracy_score
from sklearn.model_selection import train_test_split
iris = datasets.load_iris()
X = iris.data
y = iris.target
target_names = iris.target_names
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=1/3)
pca = PCA(n_components=2)
pca.fit(X_train)
X_train_pca = pca.transform(X_train)
X_test_pca = pca.transform(X_test)
clf = LDA(n_components=2)
clf.fit(X_train, y_train)
X_train_lda = clf.transform(X_train)
X_test_lda = clf.transform(X_test)
mdl1 = LinearRegression()
mdl1.fit(X_train, y_train)
mdl2 = LinearRegression()
mdl2.fit(X_train_pca, y_train)
mdl3 = LinearRegression()
mdl3.fit(X_train_lda, y_train)
# 5. LR 모델로 예측하여 성능을 검토한다.
y_pred1 = np.abs(np.round(mdl1.predict(X_test)))
y_pred2 = np.abs(np.round(mdl2.predict(X_test_pca)))
y_pred3 = np.abs(np.round(mdl3.predict(X_test_lda)))
print('* 원데이터 회귀모델성능 : ', accuracy_score(y_test, y_pred1))
print('* PCA데이터 회귀모델성능 : ', accuracy_score(y_test, y_pred2))
print('* LDA데이터 회귀모델성능 : ', accuracy_score(y_test, y_pred3))* 원데이터 회귀모델성능 : 0.98
* PCA데이터 회귀모델성능 : 1.0
* LDA데이터 회귀모델성능 : 0.98
[K-Nearest Neighbor]
- 나로부터 가장 가까운 데이터 K개를 찾아서 그 데이터가 무엇인가에 따라서 내가 어디에 속하는지 알 수 있는 것
- 전체의 점을 볼 필요가 없기 때문에 학습의 개념이라고는 볼 수 없고, Lazy Learning이라고 함.
- K개의 데이터가 나와의 거리를 모두 구해야 함 → 트리구조로 탐색함
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn import decomposition
from sklearn.decomposition import PCA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
from sklearn.neighbors import KNeighborsClassifier
from sklearn.metrics import accuracy_score
# 1) IRIS 데이터셋을 가져오기
# 2) Train/Test split 한다.
# 3) Train 데이터를 PCA/LDA를 통해 적당히 차원을 축소
# 4) PCA/LDA 를 통한 데이터로 KNN 모델 학습
# 5) KNN 모델로 예측하여 성능 검토
# x : Sepal Length, Sepal Width, Petal Length, Petal Width
# y : 0 - Setosa, 1 - Versicolour, and 2 - Virginica
iris = datasets.load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=1/3)
pca = PCA(n_components=2)
pca.fit(X_train)
X_train_pca, X_test_pca = pca.transform(X_train), pca.transform(X_test)
lda = LDA(n_components=2)
lda.fit(X_train, y_train)
X_train_lda, X_test_lda = lda.transform(X_train), lda.transform(X_test)
knn = KNeighborsClassifier(n_neighbors=3)
knn_pca = KNeighborsClassifier(n_neighbors=3)
knn_lda = KNeighborsClassifier(n_neighbors=3)
knn.fit(X_train, y_train)
knn_pca.fit(X_train_pca, y_train)
knn_lda.fit(X_train_lda, y_train)
y_pred1 = knn.predict(X_test)
y_pred2 = knn_pca.predict(X_test_pca)
y_pred3 = knn_lda.predict(X_test_lda)
print('원데이터 KNN 모델성능 : ', accuracy_score(y_test, y_pred1))
print('PCA 데이터 KNN_PCA 모델성능 : ', accuracy_score(y_test, y_pred2))
print('LDA 데이터 KNN_LDA 모델성능 : ', accuracy_score(y_test, y_pred3))원데이터 KNN 모델성능 : 0.94
PCA데이터 KNN_PCA 모델성능 : 0.98
LDA데이터 KNN_LDA 모델성능 : 0.94
[Decision Tree Classifier]
결정권한이 큰 것을 맨 상단에 놓아야 트리가 단순해 짐.
결정권한이 큰 것을 판단하는 방법
1. 엔트로피 (entropy) 불확실성 : 좀 더 구분하기 용이한 방법으로 배열
2. 이득 정보 (Gain Index) : 어떤 인자를 한 클래스에 놓았을 때 한 뭉치로 될 가능성을 판단
- DecisionTreeClassifier( )
- criterion{“gini”, “entropy”, “log_loss”}, default=”gini”
- max_depth : 트리 최대 계층수
import numpy as np
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeClassifier
from sklearn.metrics import accuracy_score
# 1) IRIS 데이터 불러오기
# 2) Train / Test Data Split
# 3) DT 모델 생성
# 4) 학습
# 5) test 데이터로 검증
iris = datasets.load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X,y, test_size=1/3)
dt = DecisionTreeClassifier(criterion = 'entropy')
dt = dt.fit(X_train, y_train)
y_pred = dt.predict(X_test)
print('* Decision Tree 모델성능 : ', accuracy_score(y_test, y_pred))* Decision Tree 모델성능 : 0.98트리의 깊이층을 max_depth로 조정을 하다가 보면 어느 정도로 조정해야 할지 판단이 불가함
그래서, 앙상블 모델구조 등장
배깅, 부스팅
랜덤포레스트 → 전체 데이터에서 임의로 샘플을 뽑아서 DT를 만듬
- RandomForestClassifier(n_estimators=100)
- RF는 Feature importance를 제공함 : 상단에 올라오는 피처의 중요정도를 판단해줌
2022.6.10. Friday
[Support Vector Machine]
※ Data Lake 변화
Extract → Transform → Load ▶ Extract → Load → Transform
※ 테슬라 메인 제어기 ECU 가 3개 : 중앙집권적 아키텍처를 만들어 놓았다는 증거
타 사 자동차 메인 제어기는 상당히 많아 통합제어가 어려움
※ 현업 적용을 위해 첫번째로 해야할 일 : 문제 발굴, 문제 진단, 문제 정의
※ 인공지능 추천가 시스템
뉴럴넷의 단점을 개선하기 위해 만들어 진 것이 SVM
매번 학습할 때마다 정확도 변화가 있기 때문에
Slack Variable : 슬랙변수
중심선 : 결정경계
선형 중심선으로 나눌 수 없는 것을 차원을 높여서 mapping해서 결정경계를 만들어 주는 것
이 때 사용하는 mapping해 주는 것을 커널이라고 함, 가장 많이 사용하는 커널은 가우시안 커널임
SVC(*, C=1.0, kernel='rbf', degree=3, gamma='scale', coef0=0.0, shrinking=True, probability=False, tol=0.001, cache_size=200, class_weight=None, verbose=False, max_iter=- 1, decision_function_shape='ovr', break_ties=False, random_state=None)
주요 파라미터 조정
kernel{‘linear’, ‘poly’, ‘rbf’, ‘sigmoid’, ‘precomputed’} or callable, default=’rbf’
sigmoid를 사용하려면 뉴럴넷을 사용함
degree int, default=3
Degree of the polynomial kernel function (‘poly’). Ignored by all other kernels.
몇 차식으로 확장시켜줄 것인지 정하는 것
gamma{‘scale’, ‘auto’} or float, default=’scale’
Kernel coefficient for ‘rbf’, ‘poly’ and ‘sigmoid’.
- if gamma='scale' (default) is passed then it uses 1 / (n_features * X.var()) as value of gamma,
- if ‘auto’, uses 1 / n_features.
SVM은 binary classification 밖에 못한다는 한계점을 갖고 있었으나 SVM에는 다음 알고리즘이 들어 있어서 문제를 해결함, 그렇게 하여 요소가 많아도 분류할 수 있도록 하였음
- One against One (= OVO, One versus One)
- One against Rest (= OVR, One versus Rest)
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.model_selection import train_test_split
from sklearn import decomposition
from sklearn.decomposition import PCA
from sklearn.discriminant_analysis import LinearDiscriminantAnalysis as LDA
from sklearn.svm import SVC
from sklearn.metrics import accuracy_score
# 1. IRIS 데이터셋을 들고온다
# 2. Train / Test split을 한다
# 3. Train 데이터를 PCA/LDA를 통해 적당히 차원 축소
# 4. PCA/LDA를 통한 데이터로 SVM 모델 학습
# 5. SVM 모델로 예측하여 성능 검토
iris = datasets.load_iris()
X = iris.data
y = iris.target
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=1/3)
pca, lda = PCA(n_components = 2), LDA(n_components = 2)
pca.fit(X_train)
lda.fit(X_train, y_train)
X_train_pca, X_test_pca= pca.transform(X_train), pca.transform(X_test)
X_train_lda, X_test_lda = lda.transform(X_train), lda.transform(X_test)
svm_clf1,svm_clf2,svm_clf3 = SVC(),SVC(),SVC()
svm_clf1.fit(X_train, y_train)
svm_clf2.fit(X_train_pca, y_train)
svm_clf3.fit(X_train_lda, y_train)
y_pred1 = svm_clf1.predict(X_test)
y_pred2 = svm_clf2.predict(X_test_pca)
y_pred3 = svm_clf3.predict(X_test_lda)
print('* 원데이터 SVM 모델성능 : ', accuracy_score(y_test, y_pred1))
print('* PCA데이터 SVM 모델성능 : ', accuracy_score(y_test, y_pred2))
print('* LDA데이터 SVM 모델성능 : ', accuracy_score(y_test, y_pred3))* 원데이터 SVM 모델성능 : 0.88
* PCA데이터 SVM 모델성능 : 0.92
* LDA데이터 SVM 모델성능 : 0.98
[Perceptron] 인공신경망
수상돌기 : 감각기관에서 들어오는 자극을 담당하는 신경세포
수상돌기의 핵
축색돌기 : 역치 개념 존재
각각의 신경세포가 각각의 자극을 센싱하는 기능을 담당하고,
역치를 넘어서는 입력이 들어 왔는가 아닌가에 대한 ON/OFF 스위치 판단
1. 자극 : 입력 데이터 ( input(t) )
2-1. 아무런 자극이 없을 때 들어오는 정기적인 신호 : 세타 웨이트 신호
2-2. 자극이 있을 때 들어오는 신호 : 바이어스 신호
3. 활성화 함수 (가중치 전달함수)
4. 반응 : 출력 데이터 ( output(t) )
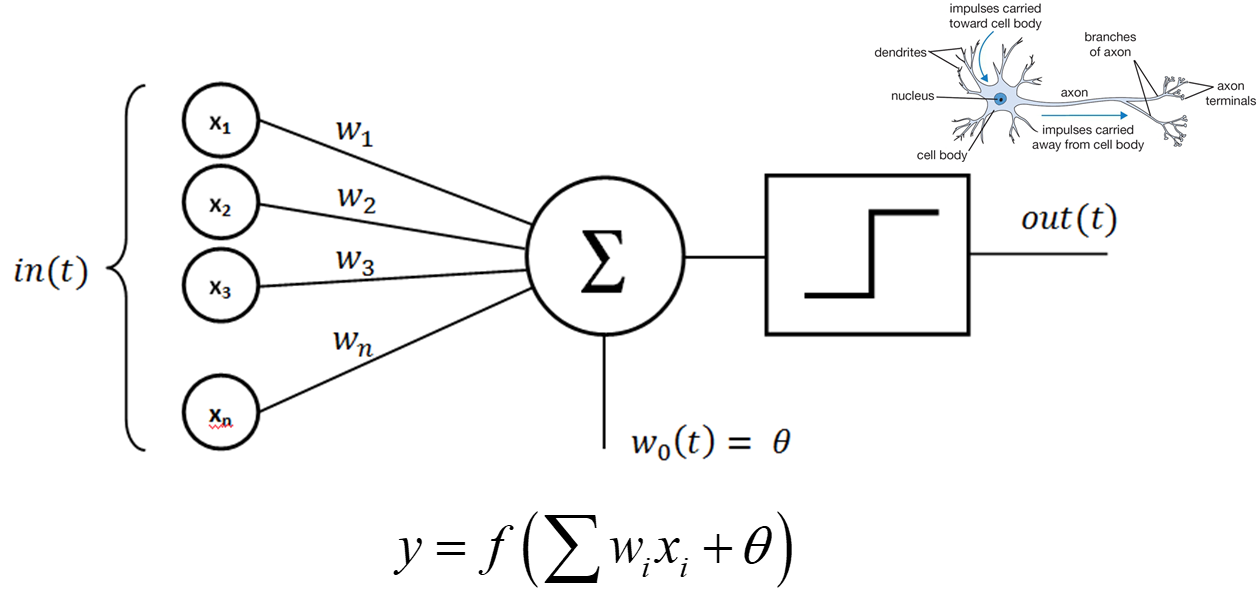
입력 데이터에 대해서 '웨이트'와 '바이어스' 신호를 학습을 통하여 식으로 만들고, 전달함수로 판단하여 출력을 보냄
y = Wx + b -> 학습된 식을 전달함수에 입력하여 출력함
W : Weight
b : Bias
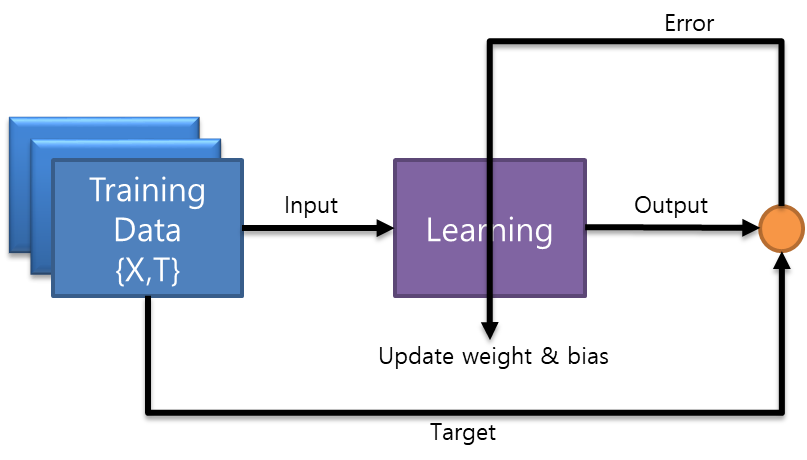
1. initialize network weight and bias : 웨이트와 바이어를 임의로 정해서 넣음
2. compute error value between target and network output : 목표값과 출력결과의 차이를 계산
3. Learning to minimize error : 차이를 최소화하는 쪽으로 학습해 나감
4. Repeat 2~3 until error becomes network goal : 목표 달성까지 계속 반복함
- Delta Rule : Weight 학습하여 변화하는 룰
어떤 입력 노드가 출력 노드의 오차에 기여했다면, 두 노드 사이의 가중치(weight)를 입력 노드의 입력과 오차에 비례하여 조절, 웨이트는 입력데이터에 대한 오차에 비례하여 조절

- 바이어스의 학습
바이어스도 마찬가지로 오차에 비례하여 조절, 바이어스는 입력데이터가 없고 1에 오차에 비례한 값을 곱하여 조절

※ model. Sequential()
※ model.add() : 모델을 한 층씩 쌓아갈 수 있게 더하는 역할
※ layers.Dense() : 덴스 레이어를 사용
- unit : 입력되는 데이터의 경로 노드수
- activation : 활성함수 종류
- input_shape : 행렬 사이즈 형태, 튜플형태 입력
※ compile()
- optimizer
- loss
- metrics
※ fit()
- epochs : 돌리는 횟수
- batch_size :
- verbose :

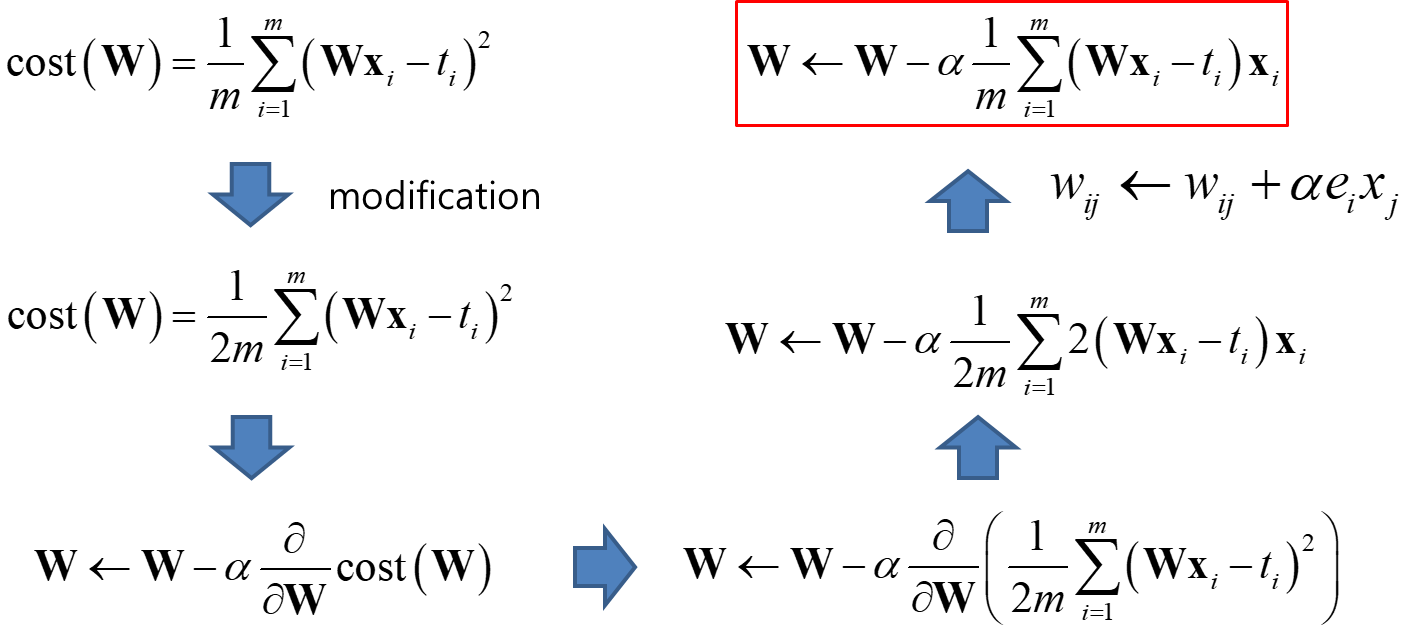
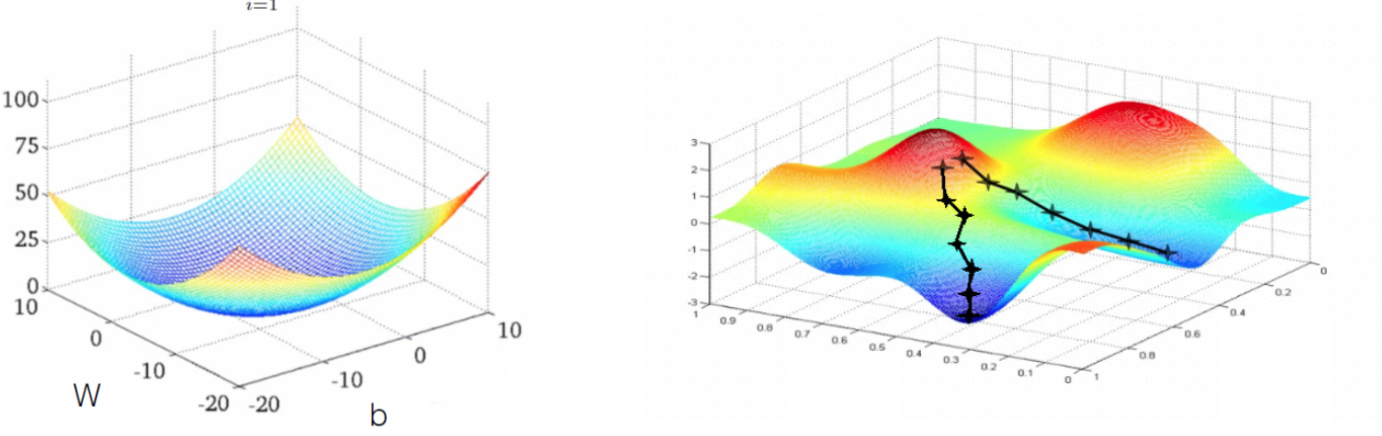
Cost Function : Loss
cost (W,b) = 에러 e (결과y값에서 타겟값 t를 빼줌)의 제곱의 평균 : Mean Square Error
신경망은 cost를 최소화하는 것이 목표임
경사하강법 (Gradient Decent) → 미분이 가능해야 하는 것이 전제
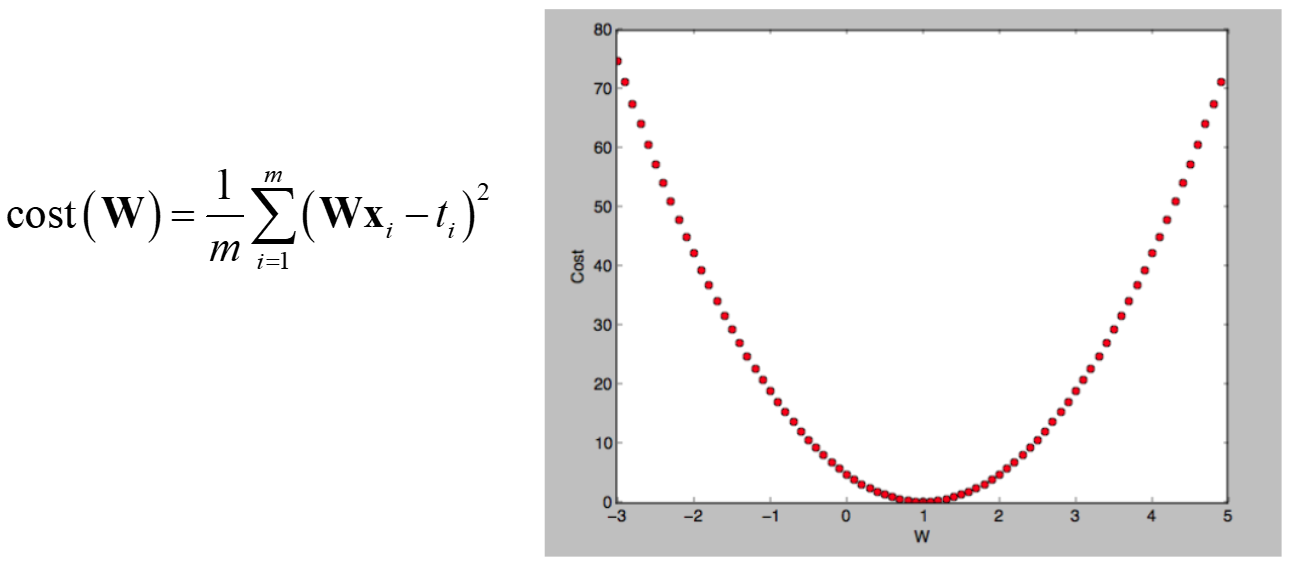
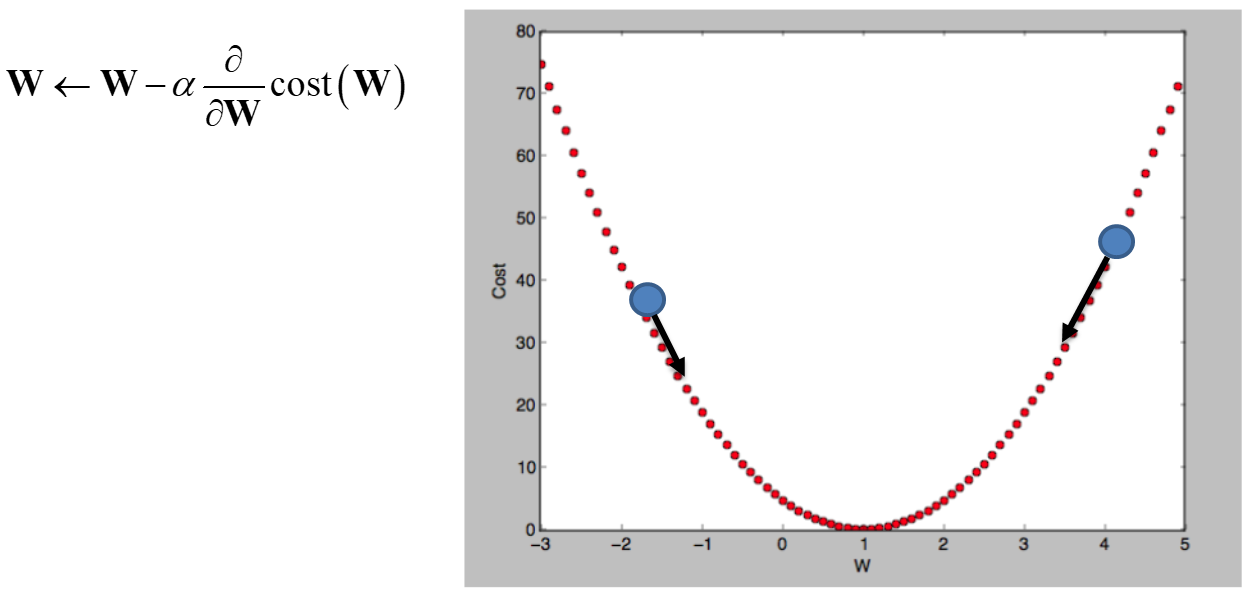
SGD
loss를 최소화하기 위해 sgd법을 통해 optimizing 하겠다는 의미, 학습이 잘 되고 있다는 것은 loss가 줄어들고 있다는 의미


SGD : Data를 GPU로 가져와서 계산하고 다시 Data를 또 가져와서 계산하는 방법, weight 갱신수는 Data의 갯수만큼 해주게 됨, batch_size = 1 일때, Data를 한 개씩 돌려보겠다는 의미, 세밀해서 정확도가 높음, 데이터량이 적을 경우.
BGD : Data를 모두 받아서 weight 갱신을 한 번만 하겠다는 것임, batch_size = 60,000라면 데이터를 한 번에 다 올려서 학습하겠다는 의미, 데이터량이 너무 많으면 적절히 사이즈를 올려서 하는 것이 좋음, batch_size와 epochs를 적절히 조절해야 나가야 함.
Mini BGD : SGD와 BGD 기능을 적절히 통합함
BGD

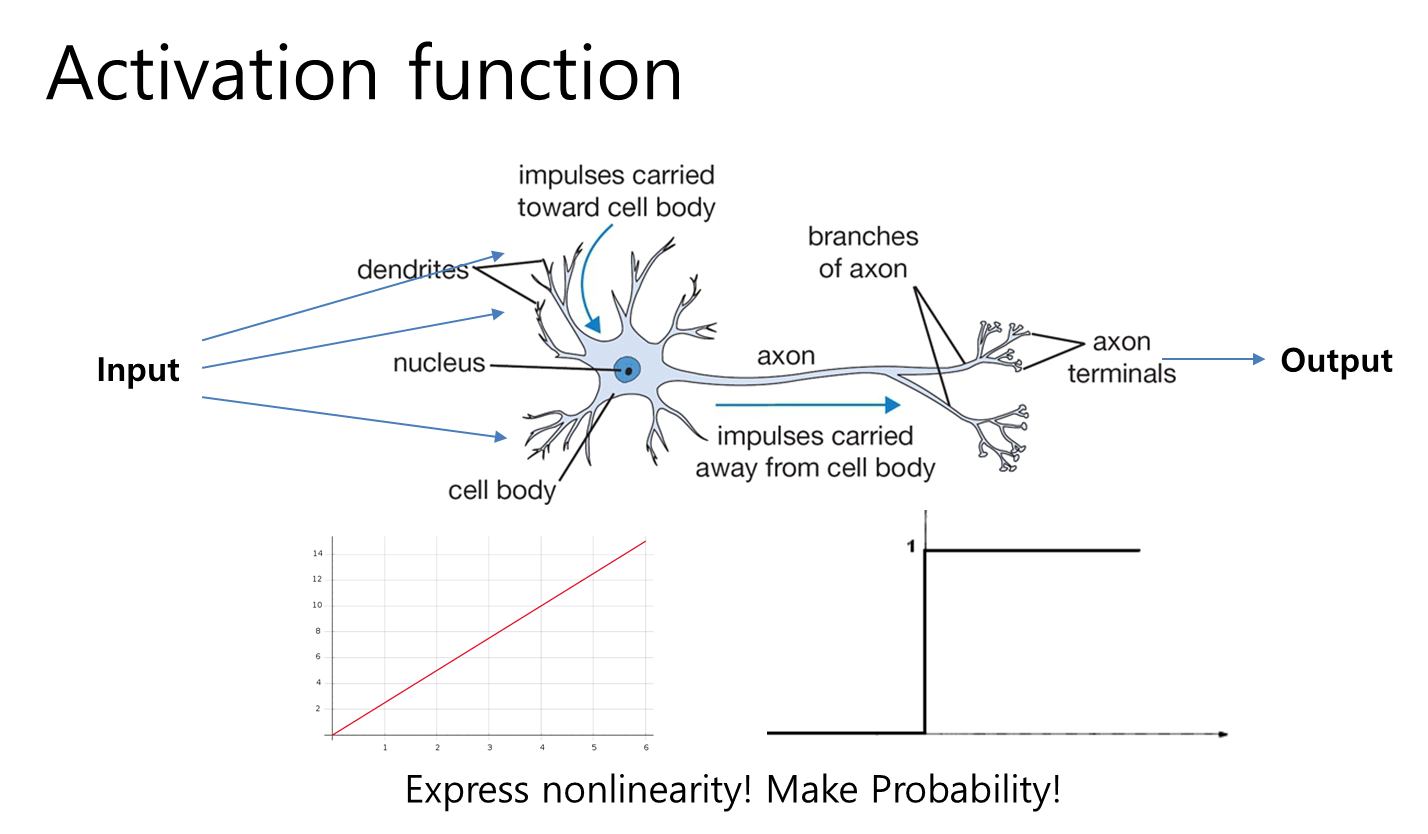
전달함수 (활성화함수)
신경망 결과를 전달할지 말지에 대한 스위치의 의미
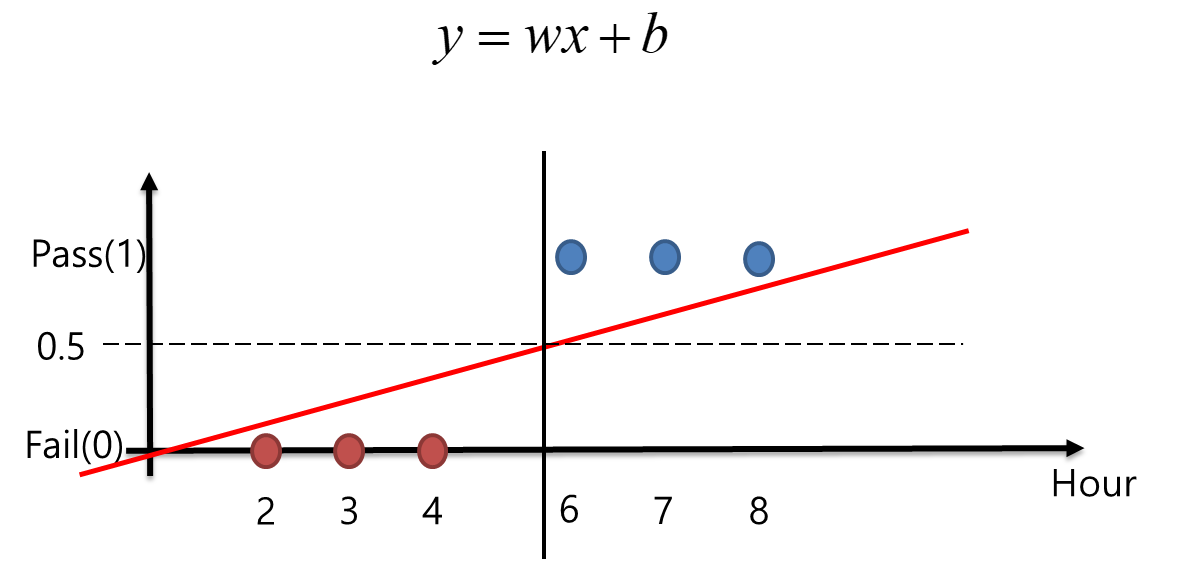
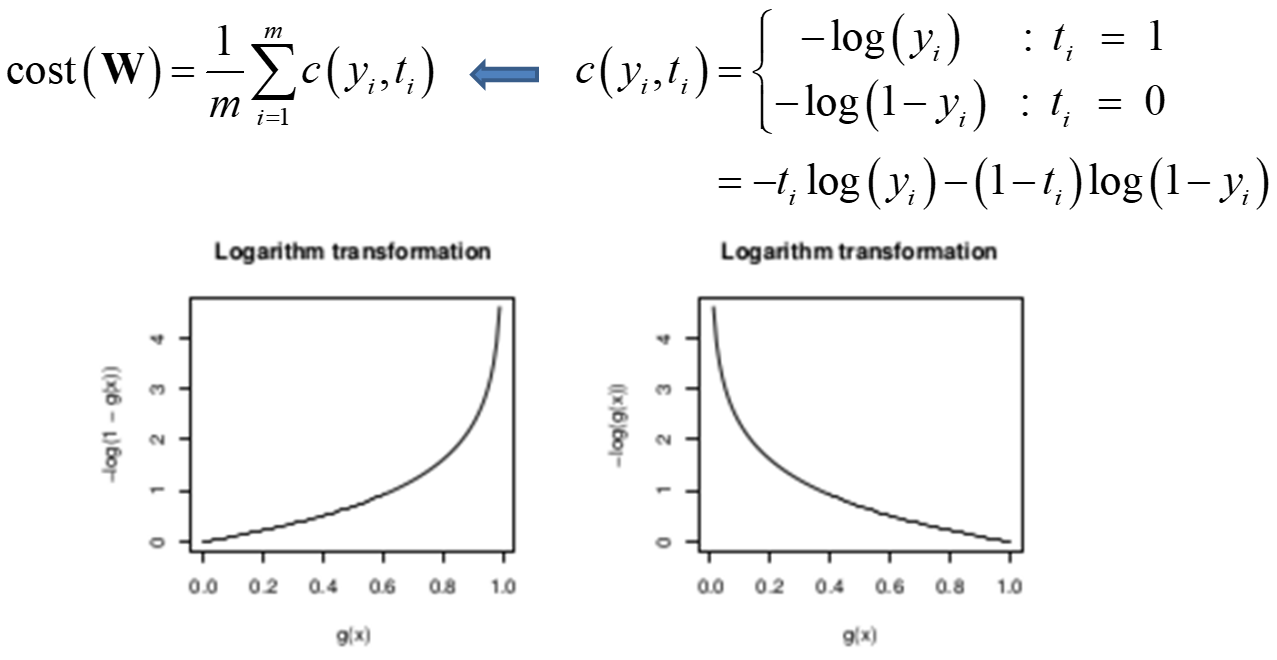
고양이냐 강아지냐 라는 판단하는 학습결과에 대한 전달함수는 sigmoid function을 사용
sigmoid function은 결과값이 확률로서 표현되게 됨.
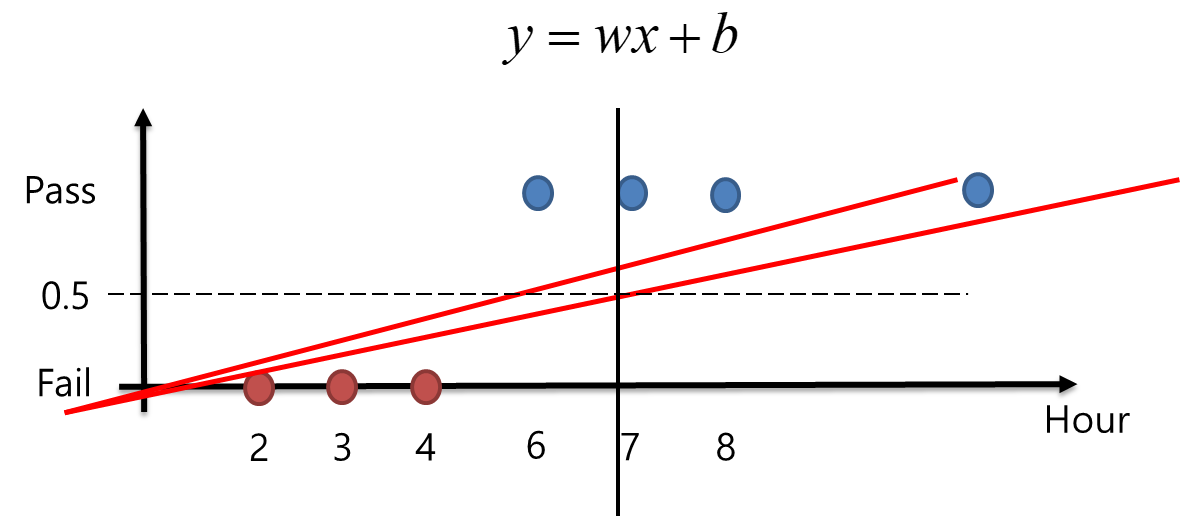


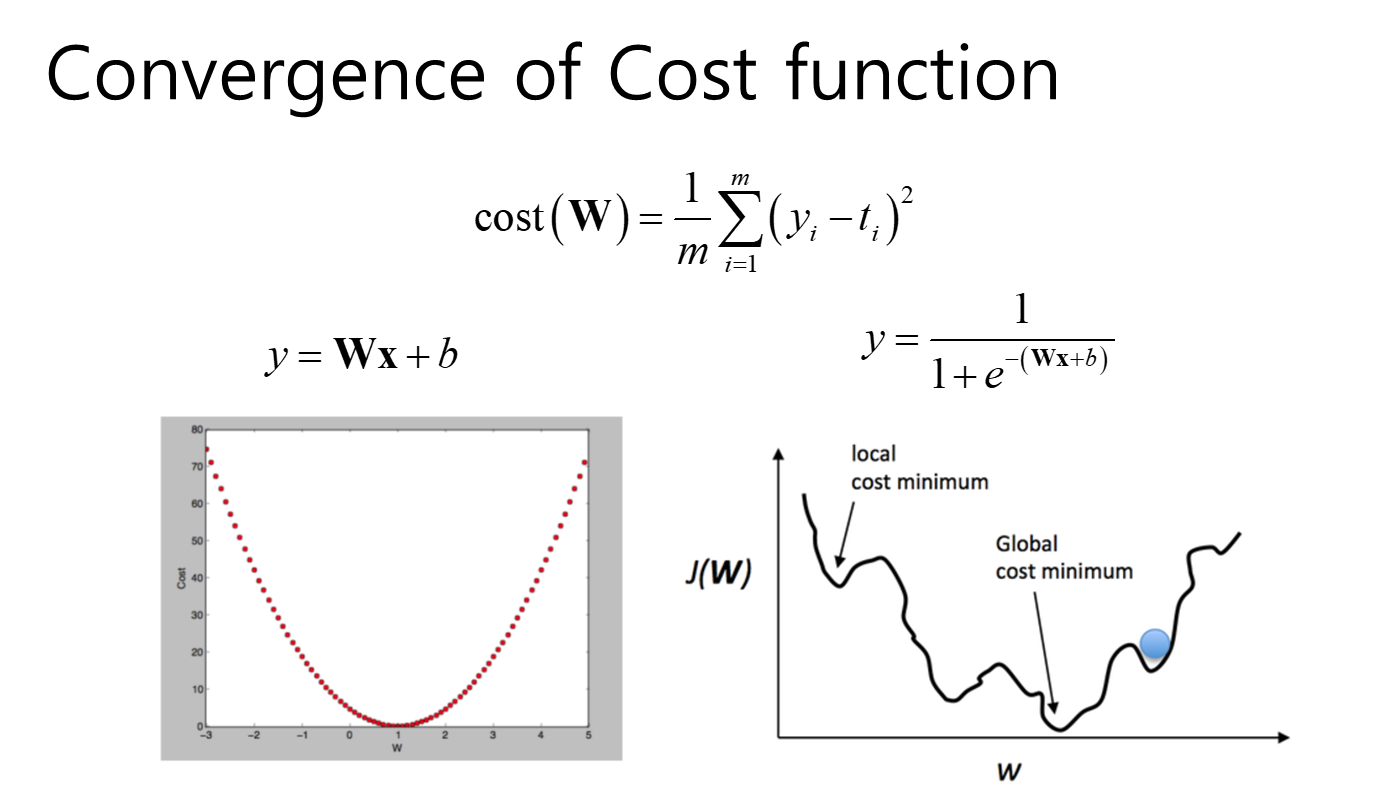
Cost Function 수렴
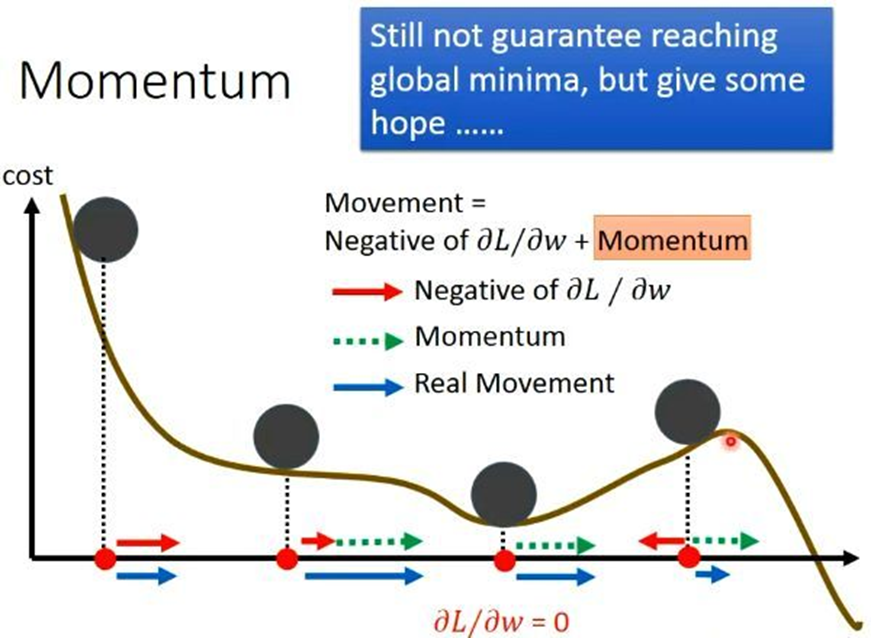
다층 신경망의 노드가 많으면 cost function이 복잡해 진다. 그래서 국소적인 최소값(local cost minimum)에 갇히는 것을 방지하기 위해서 learning rate를 조절하면서 학습시켜야 함. Adam (Adaptive Momentum)과 같이 learning rate를 관성의 힘의 원리를 이용하여 경사하강 움직임을 조정할 수가 있음.
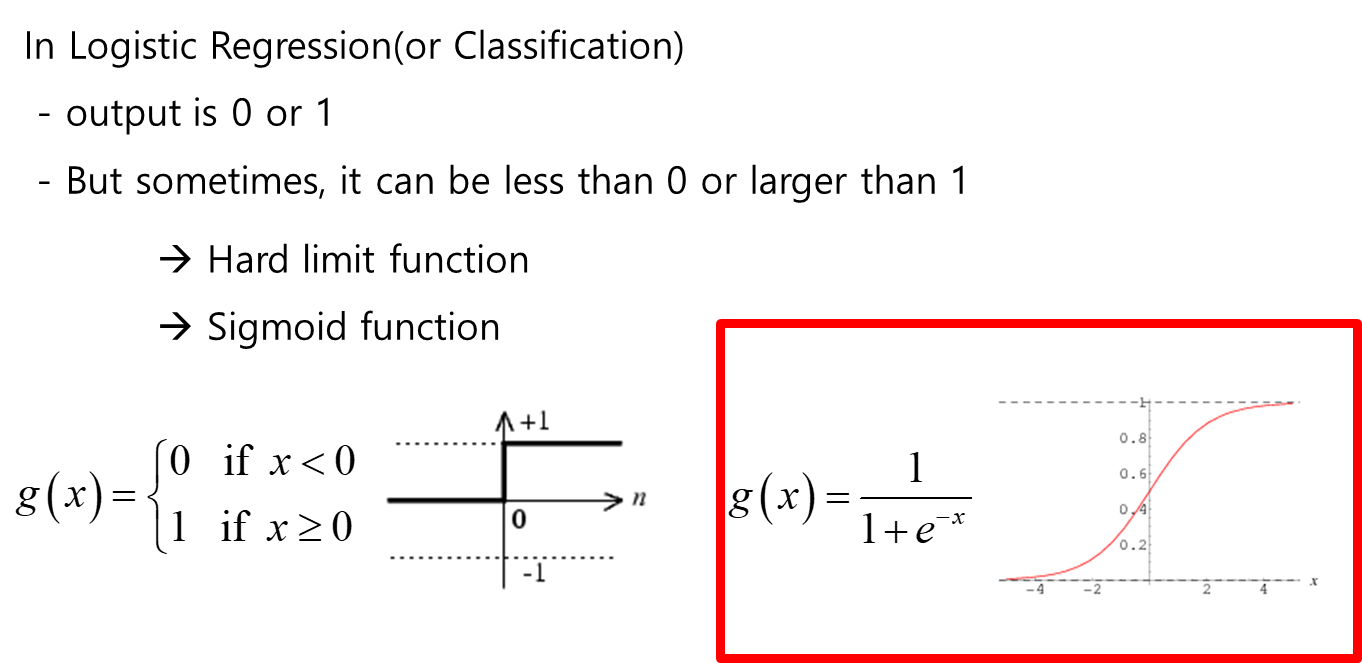
Sigmoid Function
Binary Cross Entropy Function : local minimum을 줄여주는 역할을 함
Adam
다층 퍼셉트론
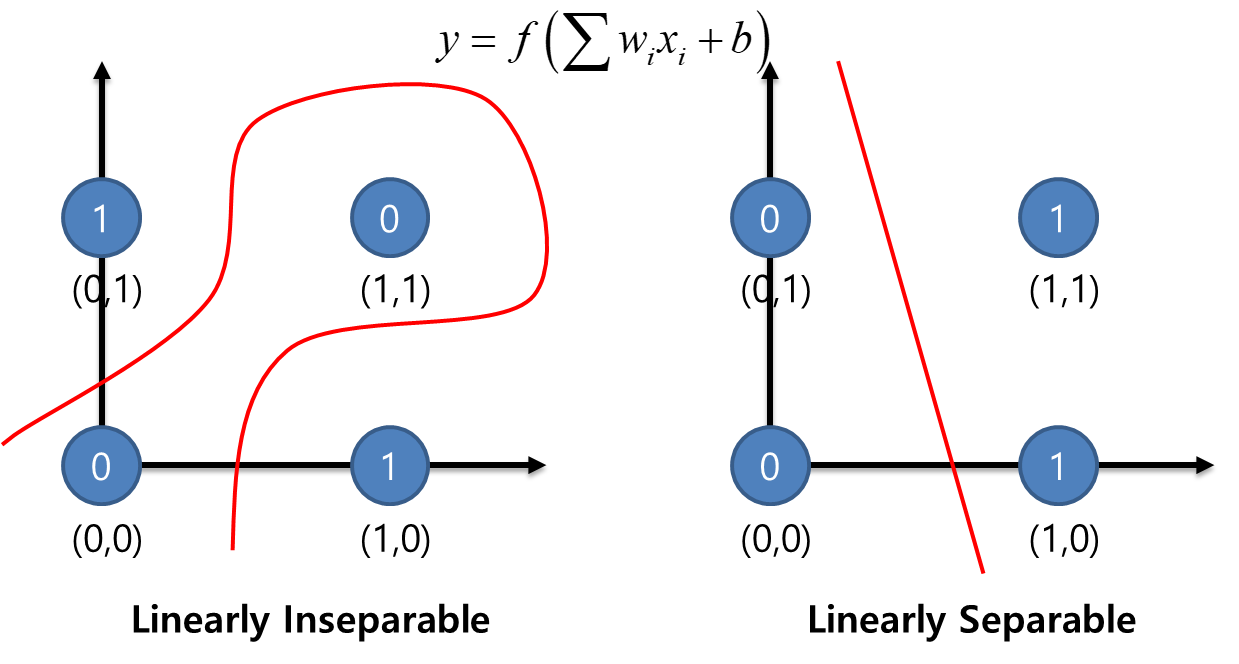
- Single layer perceptron can be applied only simple classification/regression problem
- Hyper plane should be more complex for real world model → It cause first neural network winter!
Exclusive OR : Linearly Inseparable
Exclusive NOR : Linearly Separable
Perceptron 여러 개를 가지고 Exclusive OR Gate, Exclusive NOR Gate를 표현하려고 했음
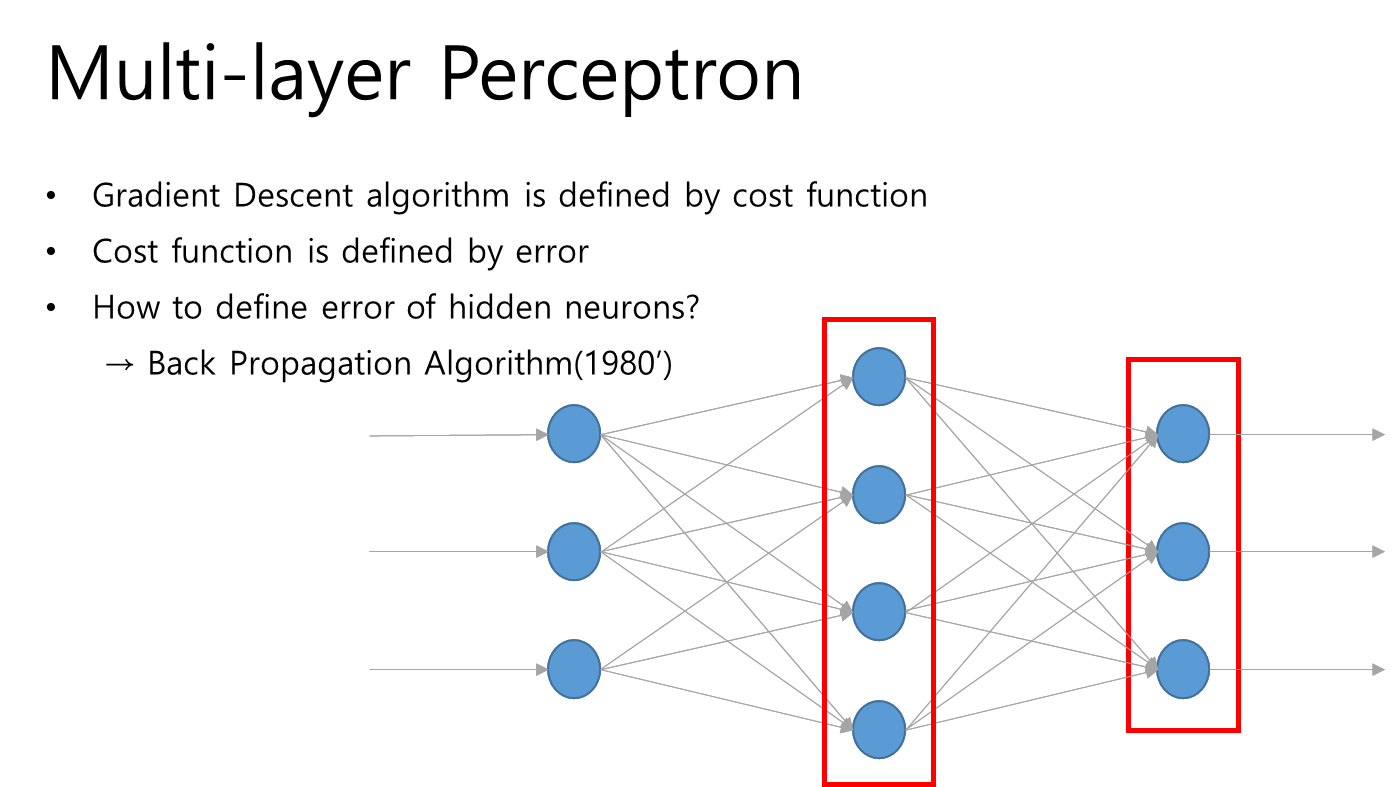
레이어 중간에서의 error와 target을 판단할 수가 없는 문제가 있었음, 즉 중간 레이어의 error값을 계산할 수가 없었기 때문에 다층 레이어 학습이 불가능했었기 때문에 20년 동안 다층 퍼셉트론은 정체되게 되었음
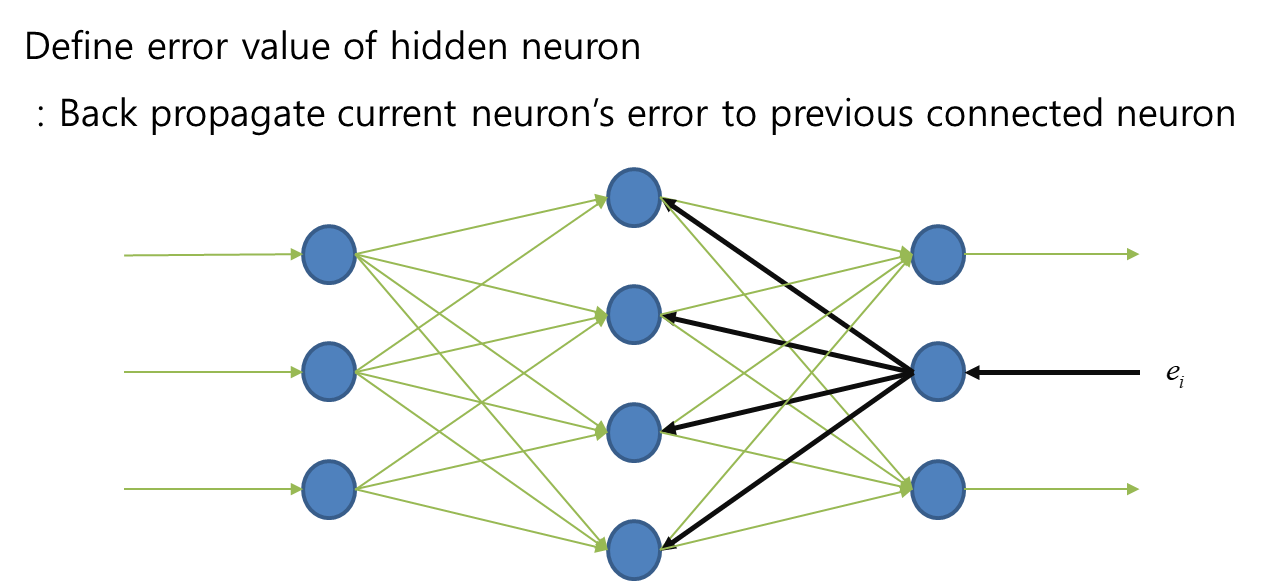
그리하여 최종 error를 중간층에서 나눠 갖자는 아이디어가 등장함, 이것이 역전파로 에러를 전달하는 원리이다.
위의 다층 구조를 다음과 같이 표현이 된다.
from tensorflow.keras import models, layers, Input, Model
import numpy as np
X = np.array([[0, 0],
[0, 1],
[1, 0],
[1, 1]])
T = np.array([0,0,0,1])
model = models.Sequential()
# input_shape : 첫층의 입력노드갯수
model.add(layers.Dense(units = 4, activation = 'sigmoid', input_shape = (2,))) # 중간층
model.add(layers.Dense(units = 1, activation = 'sigmoid')) # 마지막층
model.compile(optimizer='adam',
loss='binary_crossentropy',
metrics = ['acc'])
model.fit(X,T,epochs = 15000, batch_size=4, verbose=0)
Xtest_loss,Xtest_acc = model.evaluate(X,T)
print(Xtest_acc)
Ttest = model.predict(X)
print(Ttest)model.weights : 층마다 발생한 weight값을 알아볼 수 있음
modle.variables : 층마다 발생한 바이어스값도 볼 수 있음
EM기법 : Expectation-Maximization
일반적으로 노드가 많으면 많을수록 성능이 좋을 것이라는 것이 그래디언트 배니싱 문제로 착각에 봉착하게 됨.
그래디언트 배니싱 문제 : 다층 레이어의 중간 레이어에 그래디언트 에러가 역전파 전달되기 때문에 그래디언트는 에러의 미분값이기 때문에 전달함수도 미분값으로 전달되면 전달함수가 0, 1의 값을 제대로 전달하지 못하는 현상이 발생. 그래서 ReLU 전달함수가 등장함. Rctified Linear Unit
ReLU → 전달함수를 살리면서 에러도 역전파로 잘 전달할 수 있게 했음
Deep과 Wide의 문제발생 : 오버피팅 발생문제가 됨 → 드롭아웃 등장함
Dropout : 특정 피처를 만 개의 조건을 뽑아도 그 물체를 판단하는 것은 특정 몇 개만 만족하면 그 물체로 판단하겠다는 것
→ 드롭아웃까지 완성된 후 딥러닝 개발시장이 활성화되는 계기가 됨
구하기 쉬운 데이터로 딥러닝해 놓아서 적응시켜 놓고 계속 업그레이드해 가는 딥러닝도 계속해서 개발되게 됨

'Python' 카테고리의 다른 글
| Confusion Matrix(혼동행렬) 생성 : MNIST로 생성한 모델 예측결과 판단 (0) | 2022.07.12 |
|---|---|
| Teachable Machine을 이용한 딥러닝 모델 생성 방법 (0) | 2022.07.12 |
| 컴퓨터 비전 데이터 분석 4가지 항목 (0) | 2022.07.12 |
| MNIST 손글씨 이미지 딥러닝을 통한 숫자 이미지 분류 인식 (0) | 2022.07.05 |
| PIL과 OpenCV를 활용한 간단한 이미지 데이터 처리방법 (0) | 2022.07.04 |



